Obliczenia w próbówce
10.12.2009 - Monika Demichowicz
 
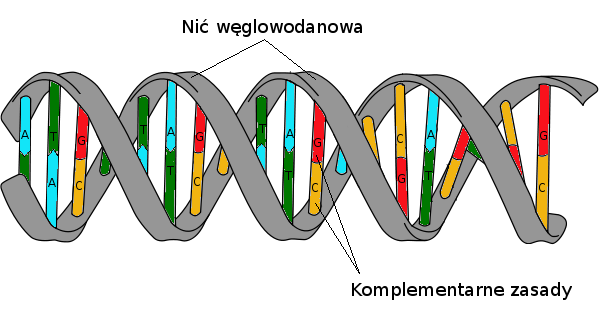
Cząsteczki, enzymy, wiązania i reakcje to chyba ostatnia rzecz, którą spodziewaliście się znaleźć na portalu informatycznym. A jednak! W tym artykule udowodnimy, że obliczenia prowadzić można również w próbówce i przeprowadzimy ciekawy eksperyment ... myślowy. Każdy, kto choć trochę interesuje się informatyką, spotkał się już zapewne z definicją algorytmu. Pokrótce, jest to sposób operowania na danych wejściowych celem osiągnięcia porządanego rezultatu. Definicja, przyznajmy, dość ogólna. I dobrze! Pewnie większości osób dane i algorytmy kojarzą się nieodmiennie z liczbami, grafami, drzewami i innymi strukturami, które zapisujemy w określony sposób w pamięci komputera. Komputera, który zasilany prądem, wykonuje programy na liczbach w zapisie binarnym. Jeśli powyższa intuicja jest Ci, drogi czytelniku, obca, nie przejmuj się - nie będziemy z niej w ogóle korzystać. W niniejszym artykule pokażemy zupełnie inne podejście do danych i obliczeń, które jednak pasuje do ogólnej definicji algorytmu. Nasz komputer będzie miał formę płynną i zmieści się w próbówce. Wszystko zaczęło się w 1994 r. od publikacji Leonarda Adlemana (tego samego, który udzielił literki A do nazwy popularnego szyfru RSA). Adleman opisał zastosowanie mechanizmów cząsteczkowych do obliczenia ścieżki Hamiltona dla pewnego przykładowego grafu. Problem Hamiltona jest popularny ze względu na swoją łatwą definicję i nietrywialne rozwiązanie. Więcej o problemie możesz przeczytać na naszym portalu w artykule "Zobacz wszystko i wracaj". Nam wystarczy prosta definicja użyta przez Adlemana: dla zdefiniowanego grafu o krawędziach skierowanych (takich, które prowadzą tylko w jedną stronę) znajdź drogę z pierwszego do ostatniego wierzchołka tak, aby przejść przez każdy z wierzchołków tylko raz. O wierzchołkach grafu można myśleć jako o miastach, które chcemy odwiedzić, a krawędzie to drogi (jednokierunkowe) między miastami. Lepkie cząsteczkiZanim przejdziemy dalej musimy poznać mechanizmy biologiczne, jakie wykorzystane zostały do rozwiązania tego problemu. Każdy, kto twierdził, że biologia i chemia nigdy mu się nie przydadzą, bo zostanie informatykiem, niech teraz wypluje swoje słowa. Zacznijmy od krótkiego przypomnienia budowy cząsteczki DNA, bo to właśnie te cząsteczki będą naszymi danymi wejściowymi. DNA jest cząsteczką zbudowaną z dwóch spiralnie skręconych nici (struktura ta zwana jest helisą). Każda z nici to nić węglowodanowa oraz szereg przytwierdzonych do niej zasad azotowych (nukleotydów).
Istnieją cztery rodzaje zasad występujących w cząsteczkach DNA: adenina - A (to A nie ma nic wspólnego z Adlemanem), cytozyna - C, guanina - G oraz tymina - T. Zasady te są parami komplementarne, czyli do siebie pasujące: A z T oraz C z G.
Oznacza to, iż zasada A z jednej nici łączy się tylko z zasadą T w drugiej nici. Analogicznie dla zasad C i G. Dlatego nici, z których zbudowane jest DNA również są komplementarne – do ich połączenia może dojść jedynie wówczas, gdy komplementarne są poszczególne pary zasad w obu niciach. Przykład dwóch nici komplementarnych: AACGCGTACGTACAAGTGTCCGAATGGCCAATG W przestrzeni cząsteczka wygląda jak poniżej. Dwie nici węglowodanowe są spiralnie skręcone, a przytwierdzone do nich zasady są parami komplementarne, co sprawia że nici do siebie pasują.
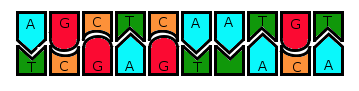
W dalszej części artykułu będziemy jednak przedstawiać DNA jako strukturę dwuwymiarową, gdyż jej przestrzenna budowa nie będzie miała znaczenia dla opisywanego przez nas eksperymentu. Zasady będziemy oznaczać różnymi kolorami, a pasujące do siebie kształty będą oznaczać komplementarność.
(3 ocen) |
Copyright © 2008-2010 Wrocławski Portal Informatyczny
design: rafalpolito.com